遗传算法求解TSP问题的DEAP实现
用GA求解N个节点的TSP问题,首先导入可能用到的库。
1 | # 导入deap相关库 |
读取数据
输入数据的组织形式根据自己组织,本文从json文件中读取TSP的城市个数和距离矩阵。
1 | # 才能够json文件中读取信息 |
问题定义
借助DEAP的Creator类可以定义算法需要的类,这些类的基类可以是Python类,如:列表list,集合set,字典dict, 也可以是PrimitiveTree等Deap内建类。基本语法是:
creator.create(name, base[,attribute[,...]]):生成名为name的类,其基类为base,具备属性attribute。示例:
1 | creator.create("Foo", list, bar=dict, spam=1) |
上述命令定义了一个名为Foo的类,等价于:
1 | class Foo(list): |
需要注意的是,尽管基类为numpy.ndarray时定义类没有什么特别需要注意的,但在复制、交叉、变异等算子中进行复制、切片时需要注意,numpy数组的切边是原数组的视图。
对于TSP问题,我们首先定义问题。下面第2行代码定义了一个名为FitnessMin的类,基类为DEAP的base模块中定义的Fitness,属性weights为目标函数的权重元组,负号表明问题为最小化。需要注意的是,weights必须为元组,哪怕为单目标优化,因此后面的逗号一定不能漏掉。
1 | # 定义问题 |
上述代码的第4行,定义了一个Individual类,具有属性fitness。其基类为list,是一个容器类,即我们在后续进化时每个个体都将存储在list中。
初始化
工具箱Toolbox提供了一系列进化算法的算子,譬如克隆函数clone可以复制个体。下面是一些常用的函数:
register函数
register(alias, method[,argument[,...]])在toolbox中注册别名为alias的函数method,如果该函数需要参数可以在注册时一并注册,也可以运行时传入。譬如:
1 | def func(a, b, c=3): |
2 3 3
1 | toolbox.myfunc(3,6) |
2 3 6
函数注册后如果要修改或重新注册,则必须先解除注册。解除注册函数为unregister(alias)。
染色体定义
TSP问题中,我们采取顺序编码,即列表中城市的编号出现顺序即为访问顺序。该初始顺序的获取可以采用random.sample抽样函数获得,并将其注册为seq函数。
1 | # random模块的sample函数:random.sample(population, k) |
完成上述注册后可以直接跟普通函数一样使用,从而得到一个初始化序列。
1 | gen1 = toolbox.seq() |
[4, 10, 5, 1, 3, 7, 12, 8, 15, 14, 6, 0, 11, 13, 9, 16, 2]
常用初始化函数
Deap的tools库提供的常用的初始化方法有:
initRepeat(container, func, n):调用func函数n次,并将结果放在容器container的一个实例中。容器container可以是列表、数组等Deap支持的容器。例如:1
2import random
tools.initRepeat(list, random.random, 2)[0.8225936649126916, 0.6002191666325332]
initIterate(container, generator):生成器generator返回的结果放在容器container的一个实例中。例如:1
2
3
4
5
6
7
8
9
10
11
12# 菲波拉契数列生成器
def fib(m):
n, a, b = 0, 0, 1
while n < m:
yield b
a, b = b, a+b
n= n + 1
from functools import partial
gen_idx = partial(fib, m=5)
a2 = tools.initIterate(list, gen_idx)
a2[1, 1, 2, 3, 5]
initCycle(container, seq_func, n=1):循环调用函数列表seq_func中的函数n次,并将结果置入容器container中。示例:1
2
3
4gen_un = partial(random.uniform, 5, 10)
func_seq = [lambda: 'a' , random.random, gen_un]
a3 = tools.initCycle(list, func_seq, 3)
a3['a', 0.8030720384393988, 7.8457022332673265, 'a', 0.5446743044675127, 8.752604169941414, 'a', 0.6014232944342702, 9.736003828310825]
定义个体
TSP问题中,每个个体只有一条染色体。注册个体生成函数individual,内容为调用函数tools.initIterate,两个参数分别为creator.Individual和toolbox.seq。根据前面initIterate介绍,函数individual实际功能就是将seq的结果放在容器Individual中。
1 | toolbox.register("individual", tools.initIterate, creator.Individual, toolbox.seq) |
1 | ind1 = toolbox.individual() |
[2, 0, 5, 3, 6, 8, 1, 12, 14, 11, 16, 13, 10, 4, 7, 9, 15]
类似的定义种群函数population
1 | # 定义种群 |
[[5, 7, 6, 12, 14, 10, 11, 15, 8, 3, 1, 0, 9, 13, 4, 2, 16], [16, 10, 9, 1, 2, 14, 5, 3, 7, 8, 6, 13, 0, 12, 4, 15, 11], [6, 4, 15, 16, 10, 1, 0, 11, 14, 8, 9, 3, 12, 13, 2, 5, 7], [13, 16, 11, 8, 15, 6, 5, 4, 1, 14, 12, 2, 7, 3, 9, 0, 10], [9, 2, 15, 10, 7, 14, 5, 0, 6, 13, 1, 3, 12, 4, 8, 16, 11]]
定义算子
遗传算法进化过程中需要进行交叉、变异和选择,DEAP内置了很多经典算子,部分如下:
| CrossOver | Mutation | Selection |
|---|---|---|
| cxOnePoint() | mutGaussian() | selTournament() |
| cxTwoPoint() | mutShuffleIndexes() | selRoulette() |
| cxUniform() | mutFlipBit() | selBest() |
| cxPartialyMachted() | mutUniformInt() | selNSGA2() |
| cxBlend() | mutESLogNormal() | selNSGA3() |
更多内置算子及其用法介绍请参考Deap官方文档。本例中我们直接使用内置的交叉、变异和选择算子。
1 | toolbox.register("mate", tools.cxPartialyMatched) |
交叉
本例中的交叉算子采用PMX交叉,交叉后返回两个子个体。
1 | ind1 = toolbox.individual() |
[2, 0, 3, 15, 8, 6, 16, 9, 14, 7, 5, 1, 13, 12, 10, 11, 4]
1 | ind2 = toolbox.individual() |
[6, 4, 16, 2, 1, 9, 10, 13, 0, 8, 3, 5, 15, 7, 14, 12, 11]
1 | # 克隆ind1,ind2 |
[2, 0, 3, 15, 8, 6, 16, 9, 14, 7, 5, 1, 13, 12, 10, 11, 4]
1 | child2 |
[6, 4, 16, 2, 1, 9, 10, 13, 0, 8, 3, 5, 15, 7, 14, 12, 11]
对child1和child2进行交叉:
1 | toolbox.mate(child1, child2) |
([15, 4, 16, 6, 1, 9, 10, 2, 14, 7, 5, 8, 13, 12, 3, 11, 0], [9, 0, 10, 15, 8, 2, 3, 13, 4, 1, 16, 5, 6, 7, 14, 12, 11])
变异
变异采用随机排序(shuffle)变异。
1 | mutant = toolbox.clone(ind1) |
[2, 0, 3, 15, 8, 6, 16, 9, 14, 7, 5, 1, 13, 12, 10, 11, 4]
1 | toolbox.mutate(mutant) |
([2, 0, 3, 15, 8, 6, 16, 9, 14, 7, 10, 1, 13, 12, 5, 11, 4],)
选择
适应度计算
首先定义适应度计算函数。
1 | # 定义适应度计算函数 |
回顾之前定义,每个个体都有一个名为fitness的属性,我们可以判断一个个体的属性值是否合法。
1 | # 判断适应度值是否合法(是否已经计算) |
False
对个体进行评估,并把评估函数返回值赋值给fitness的values属性。
1 | ind1.fitness.values = toolbox.evaluate(ind1) |
(4288.0,)
此时在判断适fitness是否合法:
1 | ind1.fitness.valid |
True
对于整个种群的个体进行适应度值计算,并赋值:
1 | # 计算种群pop的适应值函数 |
[(4145,), (3716,), (4915,), (4173,), (5147,)]
1 | for ind,fit in zip(pop, fitnesses): |
[[5, 7, 6, 12, 14, 10, 11, 15, 8, 3, 1, 0, 9, 13, 4, 2, 16], [16, 10, 9, 1, 2, 14, 5, 3, 7, 8, 6, 13, 0, 12, 4, 15, 11], [6, 4, 15, 16, 10, 1, 0, 11, 14, 8, 9, 3, 12, 13, 2, 5, 7], [13, 16, 11, 8, 15, 6, 5, 4, 1, 14, 12, 2, 7, 3, 9, 0, 10], [9, 2, 15, 10, 7, 14, 5, 0, 6, 13, 1, 3, 12, 4, 8, 16, 11]]
选择示例
本例中的选择算子采用锦标赛法进行选择。
1 | selected = toolbox.select(pop, 3) |
[[16, 10, 9, 1, 2, 14, 5, 3, 7, 8, 6, 13, 0, 12, 4, 15, 11], [16, 10, 9, 1, 2, 14, 5, 3, 7, 8, 6, 13, 0, 12, 4, 15, 11], [5, 7, 6, 12, 14, 10, 11, 15, 8, 3, 1, 0, 9, 13, 4, 2, 16]]
判断个体是否被选中:
1 | [ind in selected for ind in pop] |
[True, True, False, False, False]
进化控制主程序
完整的主程序如下,其中算法部分直接采用了algorithms库的esSimple,即GA最基本算法控制流程。
1 | def main(): |
1 | # 运行主程序 |
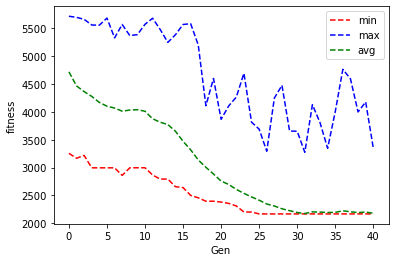
最优解为: [5, 7, 6, 0, 12, 8, 11, 15, 3, 16, 13, 14, 9, 1, 4, 10, 2] 目标函数值为: (2167.0,)
上述过程输出的收敛图如下: