进化计算框架DEAP介绍
DEAP 简介
DEAP (Distributed Evolutionary Algorithm in Python)是一个新颖的,便于快速原型开发和测试的进化计算框架。该框架试图使算法更明确,数据结构更透明。
DEAP包括如下特性:
- 可以用任何能想到数据组织方式构建遗传算法,譬如
- 列表,数组,集合,字典,树,Numpy数组等
- 进化策略(包括 CMA-ES)
- 多目标优化(NSGA-Ⅱ,NSGA-Ⅲ,SPEA2,MO-CMA-ES)
- 常见测试函数的Benckmark
- 大量示例代码
下载安装
下载安装很简单,借助pip可以快速安装。
1 | pip install deap |
使用摘要
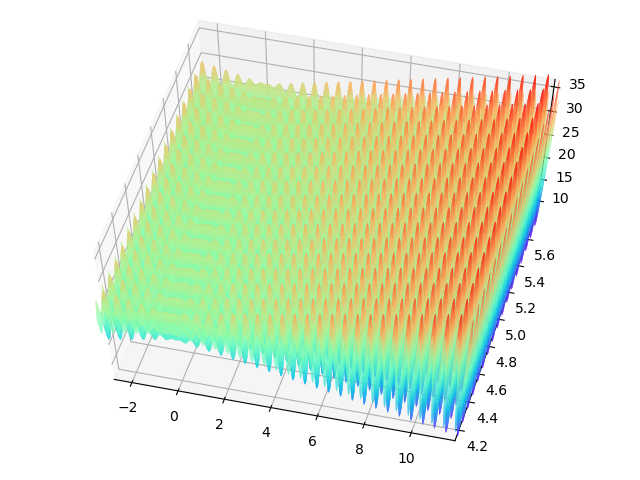
下面通过一个例子简要说明使用DEAP实现遗传算法的过程。考虑两个变量的非线性最优化求极大值问题。
\[\max f(x_1, x_2) = 21.5 + x_1 \sin(4 \pi x_1) + x_2 \sin(20 \pi x_2)\]
s.t.
\[-3.0 \leq x_1 \leq 12.1 \]
\[4.1 \leq x_2 \leq 5.8\]
这是一个多峰值函数,其图像如下图所示。
Types
使用DEAP框架实现GA之前,首先要确定问题的适合类型(是什么样的问题,最大问题,最小问题?单目标,多目标?数据如何组织,列表,还是字典?)这些都需在creator模块中实现。下面的代码中创建了一个单目标的最小化类FitnessMin和个体类Individual。
1 | from deap import base,creator |
FitnessMin类的基类为base.Fitness,属性weights为目标的权重元组,此例中只有一个元素-1.0,负值表示优化方向为最小化,只有一个权重则表示目标为单目标。Individual类的基类为list,其fitness属性为刚刚定义的FitnessMin,因此Individual具备一些列表的属性和方法。
Initialization
一旦生成了types,我们还需进一步对其初始化,即用随机值或给定值进行填充。初始化时首先要确定个体的编码方式,本例采用二进制编码。假定要求的精读为小数点后5位,则\(x_1\)需用18位二进制表示,\(x_2\)需用15位二进制数表示。DEAP提供了简易的初始化方法。
1 | import random |
上述代码中,IND_SIZE=10表示染色体长度为10。首先在toolbox中注册了方法attribute,attribute是注册名,注册内容是调用random.randint。因此可以这样理解,该方法相当于初始化染色体中的一个基因,该基因值通过random.randint(0,1)进行初始化,即为0-1随机变量。因此,直接运行toolbox.attribute就相当于运行random.randint(0,1),会返回一个0-1随机数。
接着,定义了注册individual方法,注册内容为tools.initRepeat,这是一个deap中的初始化函数,其基本语法是initRepeat(container, func, n),后面两个参数func和n表示函数func将被调用n次,而返回结果将会存贮在容器container中。本例中,将individual注册为:调用toolbox.attribute方法IND_SIZE次,并将结果存贮在Individual中(实质就是存储在列表中)。因此调用toolbox.individual就是初始化了一个个体(该个体只有一条染色体,染色体有10个基因位)。
很明显,最后一句就是注册population为初始化种群,种群的所有个体都存储在列表中。当然,此处没有指定参数n的取值,即没有指定种群规模,须在调用时指定。譬如,toolbox.population(n=50)即为初始化50个个体的种群
Operator
种群初始化后,还需注册遗传算子,譬如交叉、变异等。DEAP的toolbox模块中定义了大量常见的交叉、变异、选择算子,可以直接注册使用。
1 | def decode(ind): |
上述代码中,首先定义了适应值函数evaluate,需要特别注意的是适应函数返回的结果必须为元组(只返回一个数值时,不要漏掉后面的逗号)。然后,分别注册了交叉、变异、选择和评估函数,前三者都是直接采用自带算子:单点交叉、反转变异和锦标赛选择。
Algorithms
现在所有事情都准备好,我们可以遗传算法的迭代过程:
1 | def main(): |
上述遗传算法进化过程是标准的遗传算法,如果借助DEAP的algorithms模块,可以更简介。此外,借助Statistics模块可以更简单第地记录进化过程中的统计信息,并借助Python画图库matplotlib可以直观的展现出来。在后续更多的例子中,会介绍到更多相关库的用法和例子。
上述例子的完整代码请见我的Github。